应用聚类算法比选择最佳算法要容易得多。 每种类型都有其优缺点,如果您想要一个整洁的集群结构,就必须认真考虑。
数据聚类是安排正确的整个数据模型的重要步骤。为了进行分析,应根据共同点整理信息。 主要的问题是,什么样的公共参数提供最好的结果以及“最好”包含什么意思。
本文介绍了最广泛的聚类算法及其深入阐述。根据每种方法的特殊性,提供了对使用其应用的建议。
四种基本算法以及如何选择
根据聚类模型,可以区分四种常见的算法类别。一般而言,算法不少于100种,但是它们的流行程度以及应用领域都不是较为广泛。
基于整个数据集对象之间距离的计算,被称为基于连接的或分层的。根据算法的“方向”,它可以联合或相反地分割信息数组——聚集和分裂的名称就是从这种精确的变化中出现的。最流行或者说最合理的类型是凝聚型,您首先输入数据点的数量,然后将这些数据点合并成越来越大的集群,直到达到极限。
基于连接的集群化最突出的例子是植物分类。数据集的“树”开始于一个特定的物种,结束于一些植物“王国”,每个“王国”由更小的集群(门、类、目等)组成。
在应用了其中一种基于连接的算法之后,您将收到一个数据树状图,它将向您展示信息的结构,而不是其在集群上的明显分离。这样的特性既有好处也有坏处:算法的复杂性可能会变得过于复杂,或者根本不适用于层次结构很少甚至没有层次结构的数据集。还会出现糟糕的性能:由于大量的重复,完整的处理将花费大量时间。最重要的是无法得到精确的结构使用层次算法。
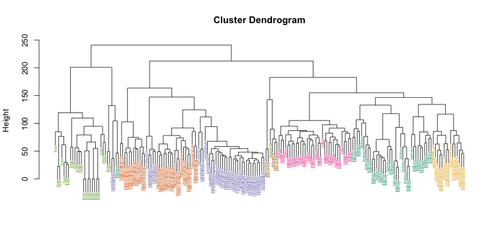
同时,需要从计数器输入的数据归结为数据点的数量,不会对最终结果产生实质性的影响,或者是预先设定的距离度量,它是粗略测量的。
根据我的经验,基于中心体的集群是最常见的模型,因为它比较简单。该模型旨在将数据集的每个对象分类到特定的集群中。集群的数量(k)是随机选择的,这可能是该方法最大的“弱点”。这种算法由于与k近邻(k-nearest neighbor, kNN)方法的相似性,在机器学习中特别受欢迎。
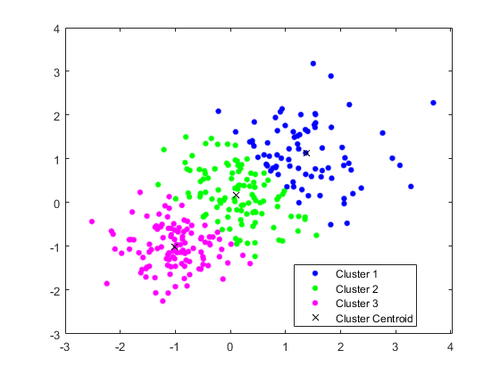
计算过程包括多个步骤。首先,选择输入数据,将数据集划分的大致聚类数。聚类的中心应放置在尽可能远的位置,这将提高结果的准确性。
其次,该算法找到数据集的每个对象与每个聚类之间的距离。最小坐标确定了将对象移动到哪个群集。
之后,将根据所有对象坐标的平均值重新计算聚类的中心。重复算法的第一步,但是重新计算了集群的新中心。除非达到某些条件,否则此类迭代将继续。例如,当集群的中心距上次迭代没有移动或移动不明显时,该算法可能会结束。
尽管数学和编码都很简单,但k均值仍有一些缺点,因此我无法在所有可能的地方使用它。那包括:
疏忽了每个集群的边缘,因为优先级设置在集群的中心,而不是边界;
无法创建一个数据集结构,该结构的对象可以按等量的方式分类到多个群集中;
需要猜测最佳k值,或者需要进行初步计算以指定此量规。
同时,期望最大化算法可以避免那些复杂情况,同时提供更高的准确性。简而言之,它计算每个数据集点与我们指定的所有聚类的关联概率。用于该聚类模型的主要“工具”是高斯混合模型(GMM),假设数据集的点通常遵循高斯分布。
k-means算法基本上是EM原理的简化版本。它们都需要手动输入集群数,这是此方法所要面对的主要问题。除此之外,计算原理(对于GMM或k均值)很简单:集群的近似范围是在每次新迭代中逐渐指定的。
与基于质心的模型不同,EM算法允许对两个或多个聚类的点进行分类-它仅向您展示每个事件的可能性,您可以使用该事件进行进一步的分析。更重要的是,每个聚类的边界组成了不同度量的椭球体,这与k均值不同,在k均值中,聚类在视觉上表示为圆形。但是,该算法对于对象不遵循高斯分布的数据集根本不起作用。这是该方法的主要缺点:它更适用于理论问题,而不是实际的测量或观察。
最后,基于数据密度的聚类成为数据科学家心中最青睐的非官方方法,包括模型的要点,将数据集划分为聚类,计数器会输入ε参数,即“邻居”距离。因此,如果对象位于ε半径的圆(球)内,则它与群集有关。
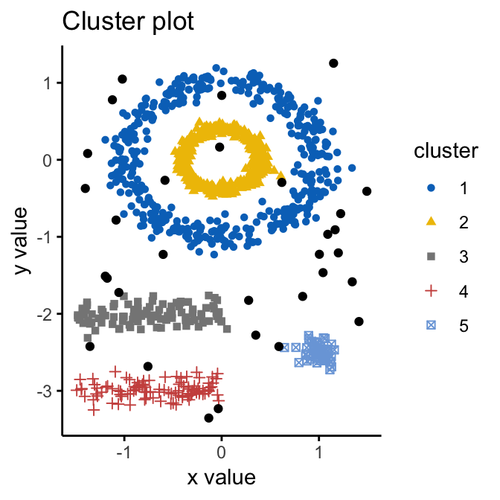
DBSCAN(基于密度的应用程序噪声空间聚类)算法会逐步检查每个对象,将其状态更改为“已查看”,将其分类到集群或噪声中,直到最后处理整个数据集。使用DBSCAN确定的集群可以具有任意形状,因此非常精确。此外,算法不会让你计算集群的数量,它是自动确定的。
不过,即使是DBSCAN这样的杰作也有缺点。如果数据集是由可变密度的数据集组成,则该方法的结果较差。如果对象的位置太近,并且无法轻松估算出ε参数,那么这也不是您的选择
综上所述,不存在错误选择的算法——它们中的一些只是更适合特定的数据集结构。为了选择最好的、更合适的算法,您需要全面了解它们的优点、缺点和特性。
有些算法可能在一开始就被排除在外,例如它们不符合数据集规范。为了避免重复的工作,你可以花一点时间来整理和记忆信息,而不是选择试错的道路。
原文作者: Josh Thompson
